即日起,分析测试中心将不定期推送冷冻电镜国内外应用案例,供各团队谋划使用冷冻电镜设备开展科研创新提供参考!
题目: Structural basis of gRNA stabilization and mRNA recognition in trypanosomal RNA editing
链接: DOI: 10.1126/science.adg4725

在转录的延伸过程中或者转录完成后,绝大多数真核生物的message RNA (mRNA) 都会经过多种RNA加工过程将mRNA前体转变为成熟mRNA,以便进行核糖体翻译。80年代中后期,研究者在锥虫 (Trypanosoma brucei) 的线粒体中首先发现了guide RNA (gRNA) 引发的RNA editing,该现象挑战了“中心法则”,揭示了遗传信息传递的新机制。这些突破性进展汇集成了gRNA引导修饰mRNA的概念,启发了后续small RNA的研究。gRNA的发现也为CRISPR-Cas的发现和技术发展奠定了基础。近三十年间,Trypanosoma RNA编辑的系统研究对共计约40种蛋白质因子进行了表征,它们构成了编辑复合体RESC (RNA-editing substrate-binding complex) 和RECC (RNA-editing catalytic complex)。其中RESC负责识别核酸底物gRNA和pre-mRNA,是整个RNA编辑的起始步骤,其相关研究对于揭示gRNA到mRNA的信息传递以及后续RNA编辑的分子机理至关重要。作为RNA editing领域的研究热点,RESC具有很强的组分不均一性和多种功能状态,这使得其机制解析具有极高的挑战性。
2023年7月6日,加州大学洛杉矶分校 (UCLA) 周正洪课题组与波士顿大学(BU) Ruslan Afasizhev课题组、上海科技大学张力烨课题组、加州大学尔湾分校 (UCI) Lan Huang课题组合作(周正洪和Ruslan Afasizhev为通讯作者;刘世恒和王宏为共同第一作者)在Science上发表题为Structural basis of gRNA stabilization and mRNA recognition in trypanosomal RNA editing的研究文章,首次解析了RNA-editing substrate-binding complex—RESC的冷冻电镜结构,阐释了多状态的RESC结合gRNA,进一步识别mRNA从而实行RNA编辑的机理。
研究者针对RESC组分高度异质性的特点,设计了多种蛋白标签并通过基因编辑将其连接到不同的RESC蛋白组分上进行内源表达和纯化测试,最终获取了组分较为均一的蛋白复合体样品。综合冷冻电镜、质谱鉴定以及cryoID等手段,解析了六个高分辨率蛋白–核酸复合体、三种不同状态RESC结构:RESC-A,RESC-B和RESC-C。
gRNA是如何被编辑体RESC识别的?
前期的分子生物学数据表明同源蛋白RESC1和RESC2可以形成多聚体,是稳定gRNA和构成RESC的核心要素,但具体的分子机制仍然未知。本研究通过结构分析以及进一步的RNA测序、蛋白突变和体内功能实验表明RESC1–RESC2二聚体参与构成了RESC-A复合体,且仅RESC2具有结合gRNA 5′端三磷酸化的关键氨基酸 (K311-R402-R424),能够识别该位点;RESC-A复合体还包含有RESC3、RESC4、RESC5和RESC6,其中RESC5–RESC6蛋白负责结合gRNA 3′端多聚尿苷化尾巴。以上结合方式促使gRNA形成“发卡状” (“hairpin-like”) 结构,可以保护其末端免于核酸酶降解,同时gRNA的“锚定”区 (“anchor” region) 和“引导”区 (guiding region) 形成了RNA茎环 (stem-loop) 结构,阻止了gRNA与mRNA的结合。
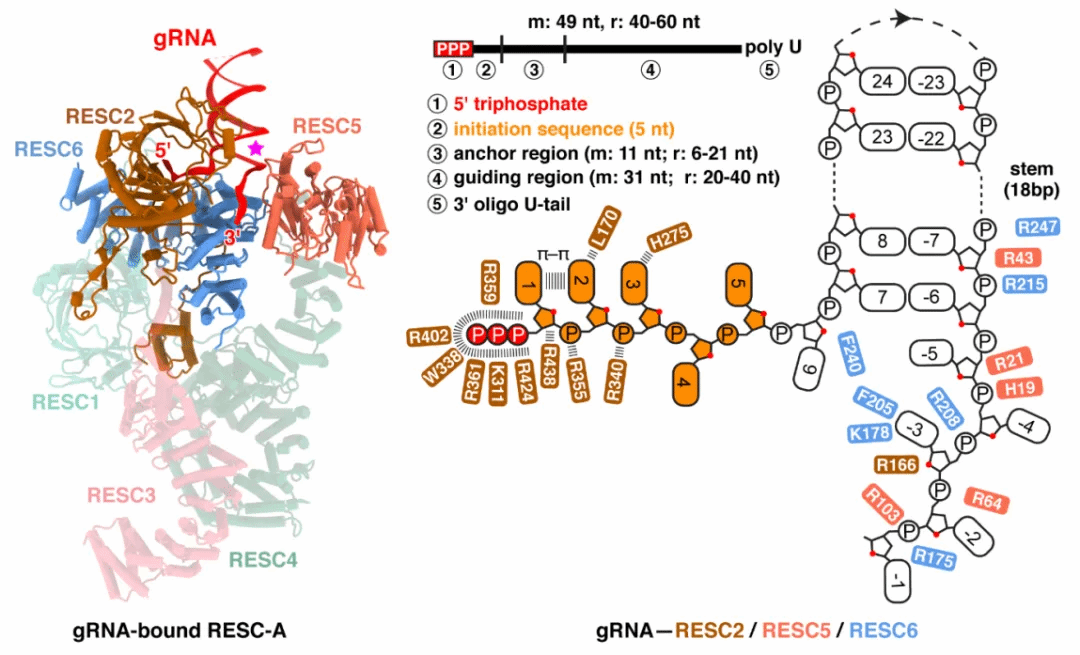
图1. RESC-A结合gRNA并将其稳定于闭合状态
gRNA如何定义mRNA编辑位点?
该过程与我们观测到的另一RESC状态—RESC-B密切相关。在RESC-B中,gRNA的5′端与RESC2脱离,其3′端仍结合于RESC5–RESC6 (亦是RESC-A和RESC-B唯一的共享蛋白组分)。该结合方式将“缺少信息的”gRNA 3′端保留在RESC-B内,从而有利于真正“富含信息的”gRNA 5′端与mRNA结合(图2A)。
与gRNA在RESC-B中的走向相反,一段长约20个核苷酸的mRNA 5′端单链和所有的RESC-B蛋白组分互作,但不与gRNA结合。此情况有效降低了gRNA与mRNA的错配,促进了mRNA 3′端与gRNA 5′端“锚定”—“引导”区的结合,进而有利于产生可供编辑的mRNA突起 (bulges) 或者环状 (loop) 位点(图2B)。
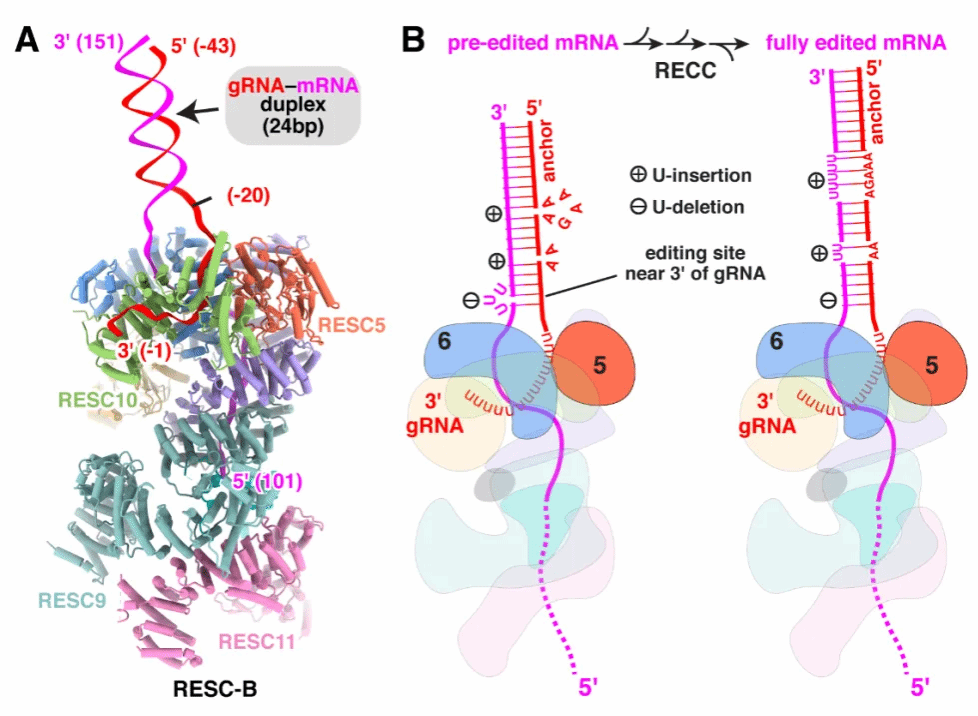
在RESC-A中,闭合的gRNA“发卡”结构表明gRNA处于“存储”状态, 暗示了该gRNA需要经历重构过程才能将其转变为mRNA可识别的单链状态。推测该过程是在RNA解旋酶复合体kREH2C的作用下,经由中间态RESC-C,使RESC-A (存储gRNA) 转变为RESC-B (gRNA识别mRNA)(图3)。在mRNA识别蛋白RESC13/12的帮助下,退火的gRNA与相应的mRNA在靠近RESC5–RESC6的区域形成不完全匹配的RNA茎 (stem),该RNA茎即可作为RECC的底物进行后续的RNA编辑。
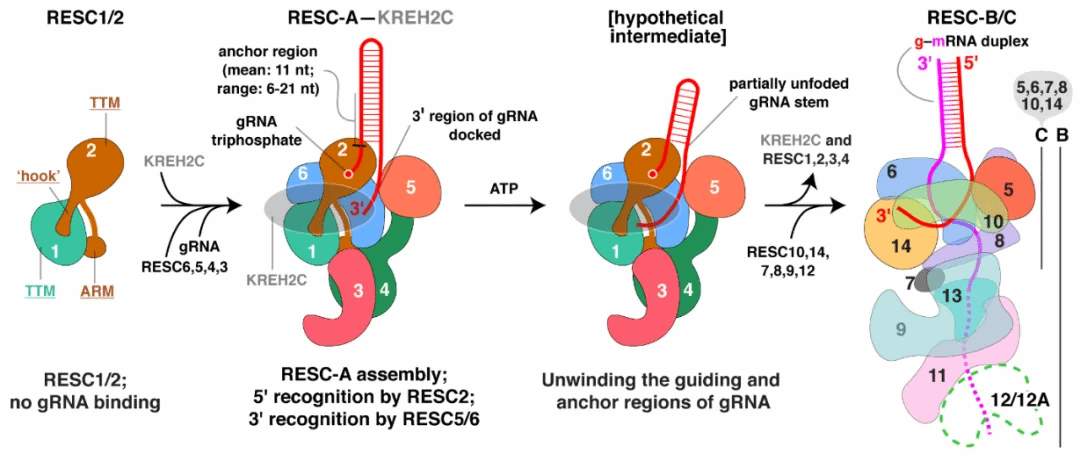
图3. RESC识别gRNA和mRNA过程中发生的状态变化
综上,该研究阐明了RESC各组分在识别gRNA和mRNA中发挥的具体功能,揭示了RESC复合体的状态变化对于底物识别和后续RNA编辑发挥关键作用的分子机制,为抗锥虫药物的研发以及RNA编辑工具的开发提供了有力支持。
本文来源 [ BioArt微信公众号]
推文原链接:https://mp.weixin.qq.com/s/I4iZzIKM_62FIKRNpODufg
福建农林大学分析测试中心
2025年11月3日