即日起,分析测试中心将不定期推送冷冻电镜国内外应用案例,供各团队谋划使用冷冻电镜设备开展科研创新提供参考!
题目: Cryo-EM structure of the transposon-associated TnpB enzyme
链接: DOI: 10.1038/s41586-023-05933-9

2023年4月13日,日本东京大学的Osamu Nureki教授团队与张锋团队合作,在Nature发表了题为Cryo-EM structure of the transposon-associated TnpB enzyme的研究论文。研究团队在真核生物中发现了一种名为TnpB的蛋白并明确其三维结构,它可能是CRISPR-Cas12酶的前体。
2类V型CRISPR效应蛋白Cas12被认为起源于IS200/IS605超家族的转座子相关TnpB蛋白。近期研究发现,TnpB蛋白是微型RNA向导的DNA核酸酶,它与一条长链非编码RNA(ωRNA)结合,切割与ωRNA向导序列互补的双链DNA靶标。然而,TnpB的RNA向导DNA切割机制及其与Cas12酶的进化关系仍不清楚。本文报道了耐辐射异常球菌ISDra2转座子来源的TnpB蛋白(简称TnpB蛋白)与同源ωRNA及靶标DNA形成复合物的冷冻电子显微镜(冷冻电镜,cryo-EM)结构。该结构显示,ωRNA采用了意想不到的构象并形成假结结构,这一假结结构在所有Cas12酶的向导RNA中均高度保守。此外,结合功能分析,该结构揭示了结构紧凑的TnpB蛋白如何识别ωRNA并切割与向导序列互补的靶标DNA。TnpB与Cas12酶的结构对比表明,CRISPR-Cas12效应蛋白通过形成不对称二聚体或插入多样的REC2结构域,获得了识别向导RNA-靶标DNA异源双链中远离原间隔序列邻近基序(PAM)一端的能力,从而能够参与CRISPR-Cas适应性免疫过程。
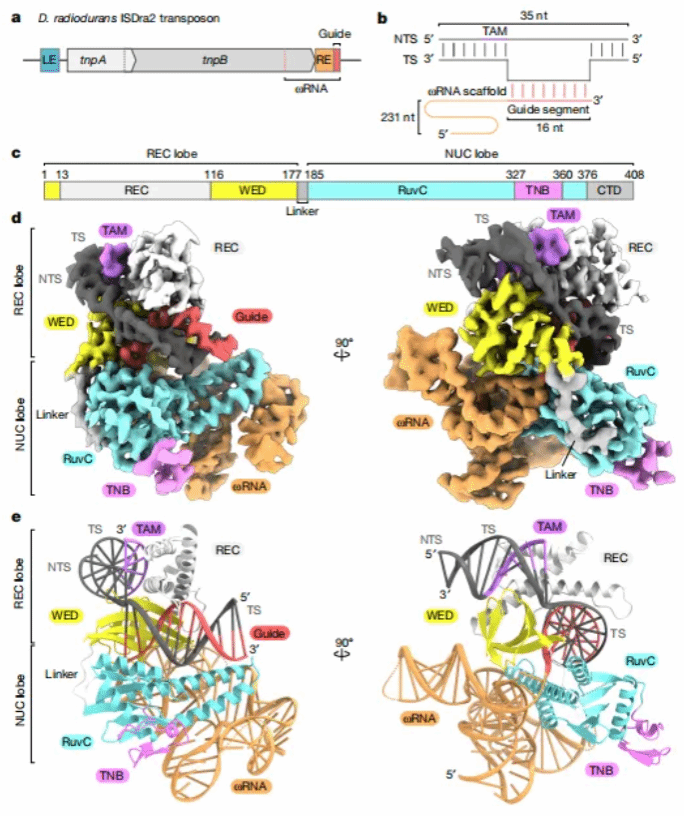
图1 TnpB–ωRNA-靶DNA三元复合体的低温电磁结构
一、TnpB–ωRNA–靶标DNA三元复合物的结构
为阐明TnpB的分子机制,作者共表达了TnpB及其同源的247核苷酸(nt)ωRNA(其3'端含16nt向导片段),随后纯化了TnpB–ωRNA复合物。通过将纯化的TnpB–ωRNA复合物与35碱基对(bp)的双链DNA(切割位点附近的DNA骨架带有硫代磷酸酯修饰,且含TTGAT靶标邻近基序(TAM))混合,重构了TnpB–ωRNA–靶标DNA三元复合物,并通过冷冻电镜(cryo-EM)分析其三元结构(图1b)。作者获得了该三元复合物的三维重构,整体分辨率为3.2Å(图1c-e)。冷冻电镜结构显示,单个TnpB分子与单个ωRNA分子组装形成核糖核蛋白效应复合物,这与先前的分子量分析结果一致。TnpB采用双叶结构,由识别叶(REC)和核酸酶叶(NUC)组成,二者通过连接环相连(图1c-e)。REC叶包含楔形结构域(WED)和REC结构域,NUC叶由RuvC核酸酶结构域和靶标核酸结合结构域(TNB)组成。C端结构域(376-408位残基)在不同TnpB蛋白中序列同源性较低,除前三个残基外,在本结构中处于无序状态。ωRNA–靶标DNA异源双链被容纳在由WED、REC和RuvC结构域形成的中央通道内(图1d,e)。含TAM的DNA双链(TAM双链)结合于WED和REC结构域形成的裂隙中,而ωRNA支架结合于WED和RuvC结构域形成的表面。在复合物内部,TnpB的氨基酸以及ωRNA和靶标DNA的核苷酸在密度图中清晰可见。相比之下,作者未观察到外围区域(如C端结构域(379-408位残基)、ωRNA支架(-70至-49位核苷酸)和TAM远端DNA双链(再杂交双链))的清晰密度,表明这些区域具有灵活性。因此,TnpB的281-296位和379-408位残基、ωRNA的-231至-117位、-70至-49位、-20至-17位及13-16位核苷酸、靶标链的-8至4位及27位核苷酸,以及非靶标链的-11位及1-24位核苷酸未纳入最终模型。
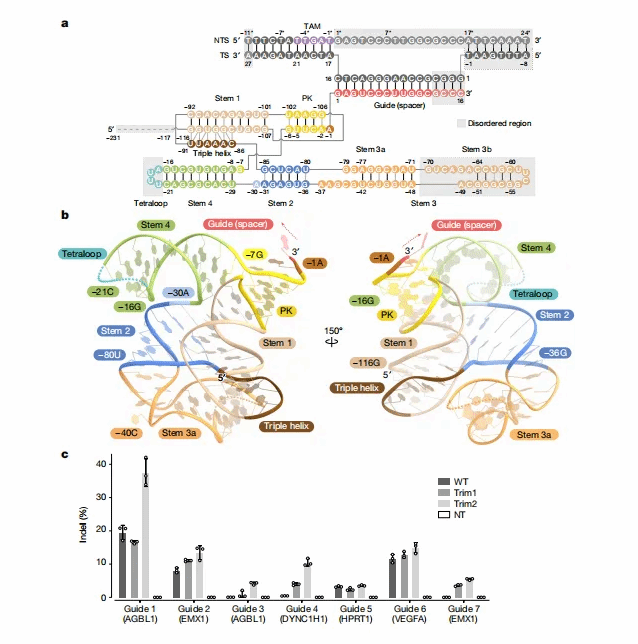
图2 ωRNA结构
二、ωRNA的结构
本结构表明,与根据一级序列预测的结构相比,TnpB–ωRNA支架采用了意想不到的结构(图2a,b)。ωRNA(-116G至16C)由16nt的向导片段和116nt的RNA支架组成,包含四个茎(茎1-4)和一个假结(PK)。值得注意的是,-5U至-3C与-103A至-105G碱基配对,而非与预测的-30A至-32G配对,且-6G和-2A分别与-102U和-106G形成非经典碱基对,从而构建假结(图2a,b及扩展数据图3c)。假结与茎1共轴堆叠,形成连续的螺旋。-35U至-32G与-81A至-84C碱基配对,且-36G和-31A分别与-80U和-85G形成非经典碱基对,进而形成茎2(图2a,b)。此外,-91U至-86C在茎1中与-111C至-116G碱基配对,从而有助于三螺旋的形成(图2a,b)。正如核苷酸序列所预期的那样,茎3a包含一个7bp的双链体(-77A至-71U与-42U至-48A配对),并带有一个环,而茎3b(-70G至-49A)未被解析,表明该区域具有内在灵活性(图2a,b)。先前的研究表明,截短Cas12fsgRNA的无序区域可提高哺乳动物细胞中的基因组编辑效率。为在TnpB中验证这一观点,作者构建了ωRNA截短突变体,其中ωRNA的5'端(-231G至-117T位核苷酸)被删除(称为Trim1)。Trim1突变体诱导插入缺失(indel)的效率与全长ωRNA相当或更高(图2c)。另一个截短突变体(通过GAAA连接子连接-71U和-48A,删除茎3b(-70G至-49A),称为Trim2)表现出进一步增强的基因组编辑活性(图2c)。这些结果证实了TnpB与Trim2ωRNA组合作为紧凑型基因组工程工具的实用性。ωRNA与Cas12酶向导RNA的结构对比显示,存在结构保守的核心区域。尽管Cas12酶的向导RNA结构不同,但它们在向导序列之前均普遍具有假结结构,而TnpB的ωRNA也形成了保守的假结结构。这些核心区域以相似的方式被其同源的TnpB和Cas12蛋白识别。ωRNA与Cas12fsgRNA的结构对比也揭示了它们之间的相似性。ωRNA的向导区、假结区和茎4区域在结构上与Cas12fsgRNA的向导区、假结区(重复-反重复1)和茎5区域(重复-反重复2)相似。此外,ωRNA的其他区域形成了与Cas12fsgRNA相似的茎环,并与TnpB蛋白相互作用。因此,作者的结构表明,ωRNA起到了“天然”sgRNA的作用,其中crRNA样区域和tracrRNA样区域通过UUUA四环(-20U至-17A)连接。
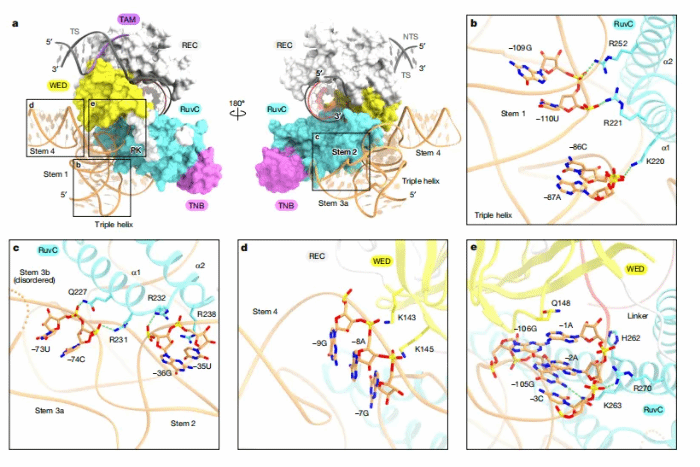
图3 ωRNA识别
三、ωRNA的识别机制
TnpB主要通过与糖磷酸骨架的相互作用,经由其WED和RuvC结构域识别ωRNA(图3a)。ωRNA的茎1和三螺旋结构通过其糖磷酸骨架与高度保守的碱性残基之间的静电相互作用,被RuvC结构域的α1和α2螺旋识别(图3b)。茎2和茎3a与RuvC结构域广泛相互作用(图3c)。-36G、-73U和-74C分别与R232、Q227和R231形成氢键相互作用,且-35U的碱基与R238形成堆叠相互作用(图3c)。茎4主要通过其上部茎区域与WED结构域的β4和β5链之间的静电相互作用,被WED结构域识别(图3d)。ωRNA的保守假结区域夹在WED和RuvC结构域之间,并被这两个结构域广泛识别(图3e)。假结中的-105G:-3C碱基对通过氢键被K263识别。假结与向导片段之间的-1A碱基夹在Q148和-106G:-2A非经典碱基对之间,且-1A的核糖部分与H262形成疏水相互作用(图3e)。值得注意的是,Cas12酶以相似的方式通过WED和RuvC结构域识别其同源向导RNA中的保守假结结构,这与ωRNA和向导RNA的核心区域在TnpB和Cas12酶中高度保守且对靶标DNA切割至关重要的观点一致。
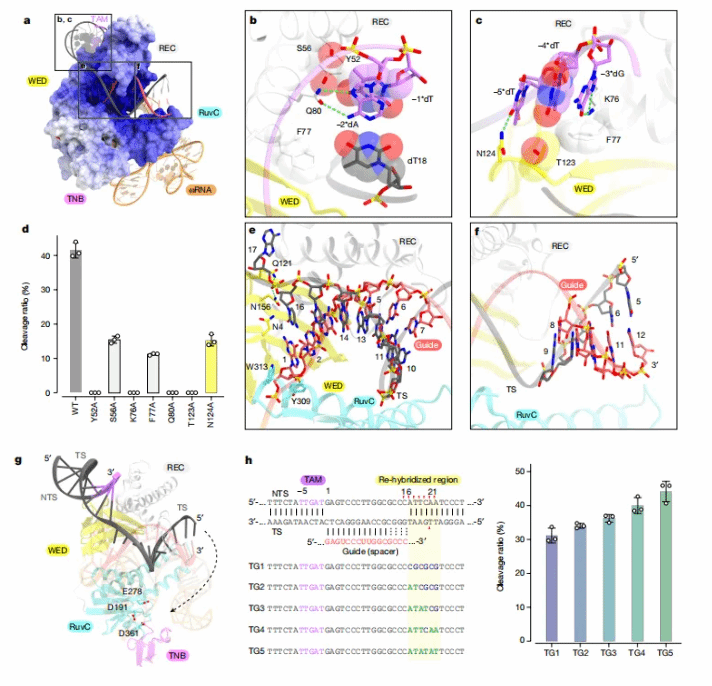
图4:靶向DNA识别与加载
四、靶标DNA的识别机制
向导RNA–靶标DNA异源双链被容纳在由REC和RuvC结构域形成的带正电中央通道内,并通过与糖磷酸骨架的相互作用被识别(图4a)。靶标链中dA17和dC16之间的骨架磷酸基团被WED结构域中的Q121和N156识别,且第一个1G:16dC碱基对分别与WED结构域中的N4和RuvC结构域中的Y309/W313堆叠(图4e),这在Cas12酶中也有观察到。这些相互作用促进了靶标DNA的解旋和向导RNA–靶标DNA异源双链的形成。由于靶标双链DNA中被置换的单链非靶标链具有灵活性,在本结构中几乎不可见。在向导–靶标异源双链中,TAM近端区域的(1G至11G):(16dC至6dC)被容纳在带正电的中央通道内,并通过其糖磷酸骨架与TnpB的静电相互作用被识别。相比之下,异源双链中的TAM远端区域(12C:3dG)暴露于溶剂中,且四个末端碱基对(13G至16C):(4dC至1dG)处于无序状态(图4f)。这些结构观察结果表明,TAM远端区域的碱基对不被TnpB识别。事实上,作者的体外切割实验表明,TAM近端的双错配(1-12位)会完全抑制TnpB介导的靶标DNA切割,而TAM远端的双错配(13-16位)会降低但仍允许TnpB切割靶标DNA。此外,作者通过在人类细胞中对TnpB进行全基因组脱靶分析,发现许多脱靶位点在TAM远端区域存在错配。这些结果表明,向导RNA–靶标DNA异源双链中约12bp的TAM近端区域对TnpB蛋白的RNA向导靶标DNA切割特异性至关重要。
Nureki团队发现与TnpB结合在一起的ωRNA具有独特的假结形状,类似于在与Cas12酶结合在一起的guideRNA中发现的假结。TnpB与Cas12酶的结构比较表明,通过不对称二聚体形成或多种REC2插入,CRISPR-Cas12效应物获得了识别guideRNA-靶DNA异源双链体的原间隔-邻近基序-远端的能力,从而参与CRISPR-Cas适应性免疫。
本文来源 [饲料用酶结构生物学]
推文原链接:https://mp.wehttps://mp.weixin.qq.com/s/R7QLz08hGJWhW0XBnYscsgixin.qq.com/s/qY92b5nyyAn6LqeMpZ4AaA
福建农林大学分析测试中心
2025年12月26日